Ver el codigo fuente de la pagina en GitHub
Parámetros de Python són iguales a los parámetros largos (
--parámetro) de Terminal, si no especificado de otra manera. Banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
gget cbio 📖
Trazar mapas de calor de la genómica del cáncer utilizando datos de cBioPortal con IDs de Ensembl o nombres de genes.
Este módulo fue escrito por Sam Wagenaar.
Argumento posicional
subcommand
O bien search o plot
Subcomando search (Python: gget.cbio_search)
Buscar IDs de estudios de cBioPortal por palabra clave.
Formato de retorno: JSON (línea de comandos) o lista de cadenas (Python).
Nota: Esto no devuelve estudios con tipos de cáncer mixtos.
Argumento posicional
keywords
Lista de palabras clave separadas por espacios para buscar, por ejemplo breast lung.
Python: Pasa palabras clave como una lista de cadenas.
Subcomando plot (Python: gget.cbio_plot)
Graficar mapas de calor de genómica del cáncer utilizando datos de cBioPortal.
Formato de retorno: PNG (línea de comandos y Python).
Argumentos requeridos
-s --study_ids
Lista separada por espacios de IDs de estudios de cBioPortal, por ejemplo, msk_impact_2017 egc_msk_2023.
-g --genes
Lista separada por espacios de nombres de genes o IDs de Ensembl, por ejemplo, NOTCH3 ENSG00000108375.
Argumentos opcionales
-st --stratification
Columna por la cual estratificar los datos. Predeterminado: tissue.
Opciones:
- tissue
- cancer_type
- cancer_type_detailed
- study_id
- sample
-vt --variation_type
Tipo de variación a graficar. Predeterminado: mutation_occurrences.
Opciones:
- mutation_occurrences
- cna_nonbinary (Nota: la
stratificationdebe ser 'sample' para esta opción) - sv_occurrences
- cna_occurrences
- Consequence (Nota: la
stratificationdebe ser 'sample' para esta opción)
-f --filter
Filtrar los datos por un valor específico en una columna específica, por ejemplo, study_id:msk_impact_2017.
Python: filter=(column, value)
-dd --data_dir
Directorio para almacenar los archivos de datos. Predeterminado: ./gget_cbio_cache.
-fd --figure_dir
Directorio para las figuras de salida. Predeterminado: ./gget_cbio_figures.
-fn --filename
Nombre del archivo de salida, relativo a figure_dir. Predeterminado: auto-generado.
Python: figure_filename.
-t --title
Título para la figura de salida. Predeterminado: auto-generado.
Python: figure_title.
-dpi --dpi
DPI de la figura de salida. Predeterminado: 100.
Banderas
-q --quiet
Solo en línea de comandos. Evita que se muestre la información de progreso.
Python: Usa verbose=False para evitar que se muestre la información de progreso.
-nc --no_confirm
Solo en línea de comandos. Omitir las confirmaciones de descarga.
Python: Usa confirm_download=True para habilitar las confirmaciones de descarga.
-sh --show
Mostrar la gráfica en una ventana (automático en notebooks de Jupyter).
Ejemplos
Encontrar todos los estudios de cBioPortal con tipos de cáncer que coinciden con palabras clave específicas:
gget cbio search esophag ovary ovarian
# Python
import gget
gget.cbio_search(['esophag', 'ovary', 'ovarian'])
→ Devuelve una lista de estudios con tipos de cáncer que coinciden con las palabras clave esophag, ovary, o ovarian.
['egc_tmucih_2015', 'egc_msk_2017', ..., 'msk_spectrum_tme_2022']
Graficar un mapa de calor de ocurrencias de mutaciones para genes específicos en un estudio específico:
gget cbio plot \
-s msk_impact_2017 \
-g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
-st tissue \
-vt mutation_occurrences \
-dpi 200
# Python
import gget
gget.cbio_plot(
['msk_impact_2017'],
['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
stratification='tissue',
variation_type='mutation_occurrences',
dpi=200
)
→ Guarda un mapa de calor de ocurrencias de mutaciones para los genes especificados en el estudio especificado en ./gget_cbio_figures/Heatmap_tissue.png.
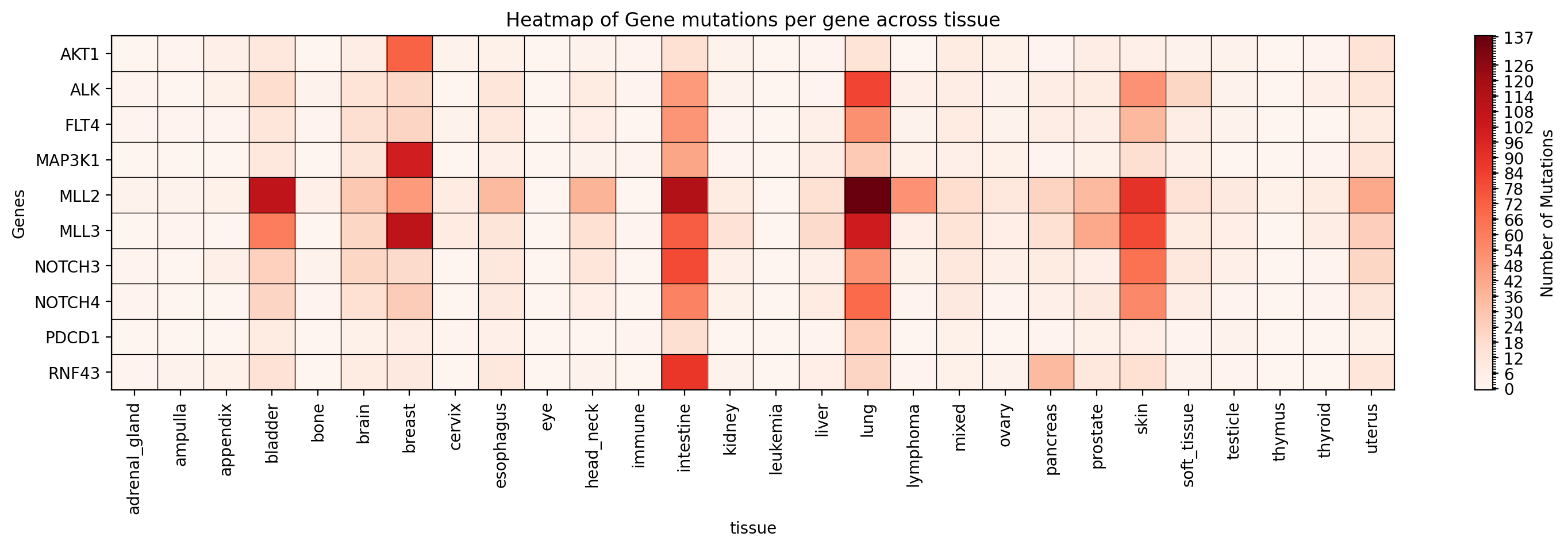
Graficar un mapa de calor de tipos de mutaciones para genes específicos en un estudio específico:
gget cbio plot \
-s msk_impact_2017 \
-g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
-st sample \
-vt Consequence \
-dpi 200
# Python
import gget
gget.cbio_plot(
['msk_impact_2017'],
['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
stratification='sample',
variation_type='Consequence',
dpi=200,
)
→ Guarda un mapa de calor de tipos de mutaciones para los genes especificados en el estudio especificado en ./gget_cbio_figures/Heatmap_sample.png.
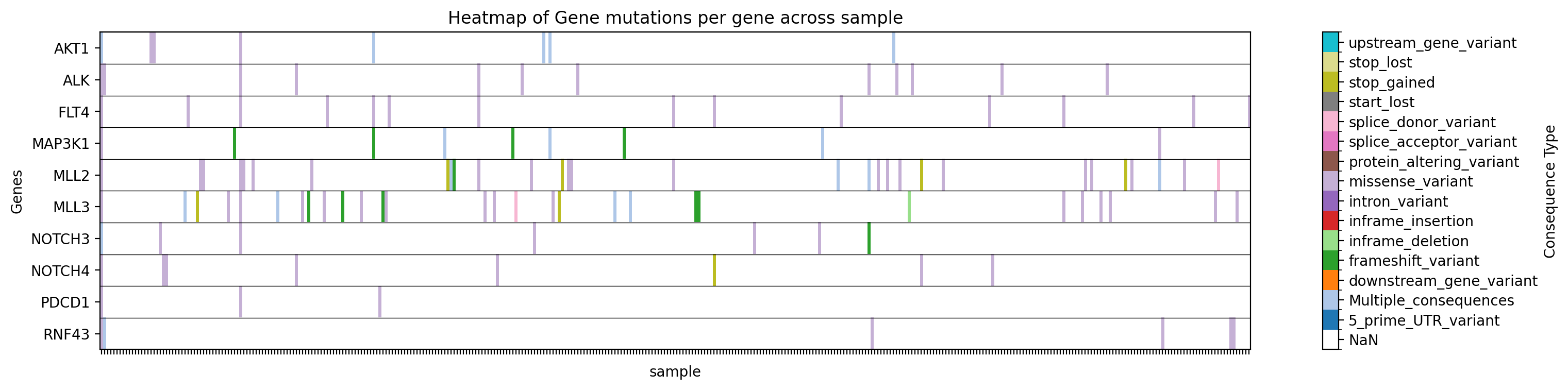
Graficar un mapa de calor de tipos de mutaciones para genes específicos en un estudio específico, filtrando por tejido::
gget cbio plot \
-s msk_impact_2017 \
-g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
-st sample \
-vt Consequence \
-f tissue:intestine \
-dpi 200
# Python
import gget
gget.cbio_plot(
['msk_impact_2017'],
['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
stratification='sample',
variation_type='Consequence',
filter=('tissue', 'intestine'),
dpi=200,
)
→ Guarda un mapa de calor de tipos de mutaciones para los genes especificados en el estudio especificado, filtrado por tejido, en ./gget_cbio_figures/Heatmap_sample_intestine.png.
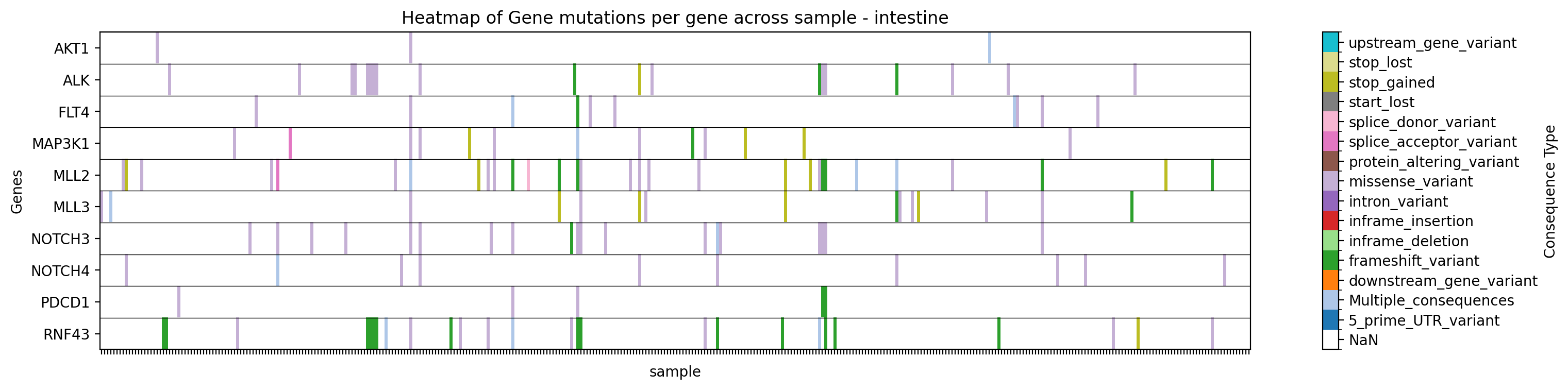
Graficar un mapa de calor con un título y nombre de archivo personalizados::
gget cbio plot \
-s msk_impact_2017 \
-g AKT1 ALK FLT4 MAP3K1 MLL2 MLL3 NOTCH3 NOTCH4 PDCD1 RNF43 \
-st sample \
-vt Consequence \
-f tissue:intestine \
-dpi 200 \
-t "Intestinal Mutations" \
-fn intestinal_mutations.png
# Python
import gget
gget.cbio_plot(
['msk_impact_2017'],
['AKT1', 'ALK', 'FLT4', 'MAP3K1', 'MLL2', 'MLL3', 'NOTCH3', 'NOTCH4', 'PDCD1', 'RNF43'],
stratification='sample',
variation_type='Consequence',
filter=('tissue', 'intestine'),
dpi=200,
figure_title='Intestinal Mutations',
figure_filename='intestinal_mutations.png'
)
→ Guarda un mapa de calor de los tipos de mutaciones para los genes especificados en el estudio especificado, filtrado por tejido, con el título "Mutaciones intestinales" en ./gget_cbio_figures/intestinal_mutations.png.
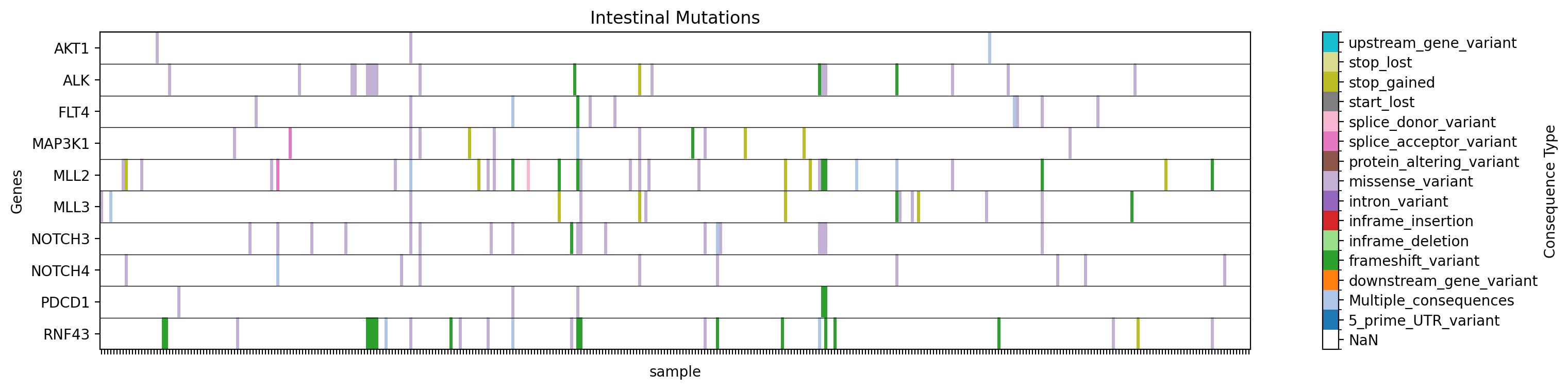
Más ejemplos
Citar
Si utiliza gget cbio en una publicación, favor de citar los siguientes artículos:
-
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
-
Cerami E, Gao J, Dogrusoz U, Gross BE, Sumer SO, Aksoy BA, Jacobsen A, Byrne CJ, Heuer ML, Larsson E, Antipin Y, Reva B, Goldberg AP, Sander C, Schultz N. The cBio cancer genomics portal: an open platform for exploring multidimensional cancer genomics data. Cancer Discov. 2012 May;2(5):401-4. doi: 10.1158/2159-8290.CD-12-0095. Erratum in: Cancer Discov. 2012 Oct;2(10):960. PMID: 22588877; PMCID: PMC3956037.
-
Gao J, Aksoy BA, Dogrusoz U, Dresdner G, Gross B, Sumer SO, Sun Y, Jacobsen A, Sinha R, Larsson E, Cerami E, Sander C, Schultz N. Integrative analysis of complex cancer genomics and clinical profiles using the cBioPortal. Sci Signal. 2013 Apr 2;6(269):pl1. doi: 10.1126/scisignal.2004088. PMID: 23550210; PMCID: PMC4160307.
-
de Bruijn I, Kundra R, Mastrogiacomo B, Tran TN, Sikina L, Mazor T, Li X, Ochoa A, Zhao G, Lai B, Abeshouse A, Baiceanu D, Ciftci E, Dogrusoz U, Dufilie A, Erkoc Z, Garcia Lara E, Fu Z, Gross B, Haynes C, Heath A, Higgins D, Jagannathan P, Kalletla K, Kumari P, Lindsay J, Lisman A, Leenknegt B, Lukasse P, Madela D, Madupuri R, van Nierop P, Plantalech O, Quach J, Resnick AC, Rodenburg SYA, Satravada BA, Schaeffer F, Sheridan R, Singh J, Sirohi R, Sumer SO, van Hagen S, Wang A, Wilson M, Zhang H, Zhu K, Rusk N, Brown S, Lavery JA, Panageas KS, Rudolph JE, LeNoue-Newton ML, Warner JL, Guo X, Hunter-Zinck H, Yu TV, Pilai S, Nichols C, Gardos SM, Philip J; AACR Project GENIE BPC Core Team, AACR Project GENIE Consortium; Kehl KL, Riely GJ, Schrag D, Lee J, Fiandalo MV, Sweeney SM, Pugh TJ, Sander C, Cerami E, Gao J, Schultz N. Analysis and Visualization of Longitudinal Genomic and Clinical Data from the AACR Project GENIE Biopharma Collaborative in cBioPortal. Cancer Res. 2023 Dec 1;83(23):3861-3867. doi: 10.1158/0008-5472.CAN-23-0816. PMID: 37668528; PMCID: PMC10690089.
-
Please also cite the source of the data if you are using a publicly available dataset.