Ver el codigo fuente de la pagina en GitHub
💡 Usuarios activos de este sitio web
Este mapa se actualiza automáticamente todos los días a las 16:00 UTC.
⬇️ Descargas diarias de gget
Este mapa se actualiza automáticamente todos los domingos a las 23:55 UTC.
🧑🤝🧑 Programas dependientes
Las siguientes aplicaciones usan gget:
- gget-mcp
- Otras proyectos que utilizan este u otros servidores MCP de
gget:
- Otras proyectos que utilizan este u otros servidores MCP de
- Biomni
Un agente de inteligencia artificial biomédica de propósito general que se está desarrollando en Stanford y Genentech. - PerTurboAgent
Un agente de auto-planificación para potenciar experimentos secuenciales de Perturb-seq. - Habilidades científicas para Claude, desarrolladas por K-Dense-AI
- Therapeutics Data Commons (TDC)
Base de inteligencia artificial para la ciencia terapéutica (código fuente, artículo en Nat Chem Bio) del laboratorio de Inteligencia Artificial para Medicina y Ciencia de Harvard. - BioDiscoveryAgent
BioDiscoveryAgent es un agente de IA basado en modelos de lenguaje para el diseño en bucle cerrado de experimentos de perturbación genética (preprint) del Proyecto de Análisis de Redes de Stanford. - DeepChopper
Modelos de lenguaje para identificar lecturas artificiales quiméricas en datos de secuenciación directa de ARN de NanoPore por el laboratorio de Yang en Northwestern. - BRAD
Un chatbot impulsado por un modelo de lenguaje para bioinformática (documentación, página principal del proyecto). - scPRINT
scPRINT es un modelo transformer grande diseñado para inferir redes génicas (conexiones entre genes que explican el perfil de expresión de la célula) a partir de datos de scRNAseq (preprint). - AnoPrimer
AnoPrimer es un paquete de Python para el diseño de cebadores en An. gambiae y An. funestus, teniendo en cuenta la variación genética en especímenes de genomas completos secuenciados de la naturaleza en datos de malariagen. - AvaTaR
Optimización de Agentes de LLM para Recuperación de Conocimiento Asistida por Herramientas (NeurIPS 2024) por el laboratorio de James Zou en la Universidad de Stanford. - GRLDrugProp
Aprendizaje de representación de grafos para modelar propiedades de fármacos. - MicrobioLink2
Analiza el impacto de la interacción huésped-microbio en la señalización posterior en células y tejidos humanos. - Implementación en Rust de gget: https://github.com/noamteyssier/ggetrs
- https://github.com/Superbio-ai/getbio
- https://github.com/yonniejon/AchillesPrediction
- https://github.com/ELELAB/cancermuts
- https://github.com/Benoitdw/SNPrimer
- https://github.com/louisjoecodes/a16z-hackathon-project
- https://github.com/EvX57/BACE1-Drug-Discovery
- https://github.com/vecerkovakaterina/hidden-genes-msc
- https://github.com/vecerkovakaterina/llm_bioinfo_agent
- https://github.com/greedjar74/upstage_AI_Lab
- https://github.com/alphavector/all
Ver también: https://github.com/pachterlab/gget/network/dependents
📃 Publicaciones selectas
- David Bradley et al., The fitness cost of spurious phosphorylation. The EMBO Journal (2024). DOI: 10.1038/s44318-024-00200-7
- Mikael Nilsson et al., Resolving thyroid lineage cell trajectories merging into a dual endocrine gland in mammals. Nature Portfolio (en revisión) (2024). DOI: 10.21203/rs.3.rs-5278325/v1
- Avasthi P et al., Repeat expansions associated with human disease are present in diverse organisms. Arcadia (2024). DOI: 10.57844/arcadia-e367-8b55
- Ibrahim Al Rayyes et al., Single-Cell Transcriptomics Reveals the Molecular Logic Underlying Ca2+ Signaling Diversity in Human and Mouse Brain. bioRxiv (2024). DOI: 10.1101/2024.04.26.591400
- David R. Blair & Neil Risch. Dissecting the Reduced Penetrance of Putative Loss-of-Function Variants in Population-Scale Biobanks. medRxiv (2024). DOI: 10.1101/2024.09.23.24314008
- Shanmugampillai Jeyarajaguru Kabilan et al., Molecular modelling approaches for the identification of potent Sodium-Glucose Cotransporter 2 inhibitors from Boerhavia diffusa for the potential treatment of chronic kidney disease. Journal of Computer-Aided Molecular Design (en revisión) (2024). DOI: 10.21203/rs.3.rs-4520611/v1
- Joseph M Rich et al., The impact of package selection and versioning on single-cell RNA-seq analysis. bioRxiv (2024). DOI: 10.1101/2024.04.04.588111
- Sanjay C. Nagi et al., AnoPrimer: Primer Design in malaria vectors informed by range-wide genomic variation. Wellcome Open Research (2024).
- Yasmin Makki Mohialden et al., A survey of the most recent Python packages for use in biology. NeuroQuantology (2023). DOI: 10.48047/NQ.2023.21.2.NQ23029
- Kimberly Siletti et al., Transcriptomic diversity of cell types across the adult human brain. Science (2023). DOI: 10.1126/science.add7046
- Beatriz Beamud et al., Genetic determinants of host tropism in Klebsiella phages. Cell Reports (2023). DOI: 10.1016/j.celrep.2023.112048
- Nicola A. Kearns et al., Generation and molecular characterization of human pluripotent stem cell-derived pharyngeal foregut endoderm. Cell Reports (2023). DOI: 10.1016/j.devcel.2023.08.024
- Jonathan Rosenski et al., Predicting gene knockout effects from expression data. BMC Medical Genomics (2023). DOI: 10.1186/s12920-023-01446-6
- Peter Overby et al., Pharmacological or genetic inhibition of Scn9a protects beta-cells while reducing insulin secretion in type 1 diabetes. bioRxiv (2023). DOI: 10.1101/2023.06.11.544521
- Mingze Dong et al., Deep identifiable modeling of single-cell atlases enables zero-shot query of cellular states. bioRxiv (2023). DOI: 10.1101/2023.11.11.566161
📰 Noticias
- Oreate AI publicación de blog: "Exploring AlphaFold2: A Guide to GitHub Repositories and Colab Resources"
gget opentargetspublicación de la plataforma Open Targets- Documental corto sobre gget: https://youtu.be/cVR0k6Mt97o
- Episodio de podcast para el Prototype Fund Public Interest Podcast sobre la importancia del software de código abierto y su papel en la investigación académica (en alemán): https://public-interest-podcast.podigee.io/33-pips4e4
- Anuncio del Prototype Fund: https://prototypefund.de/project/gget-genomische-datenbanken
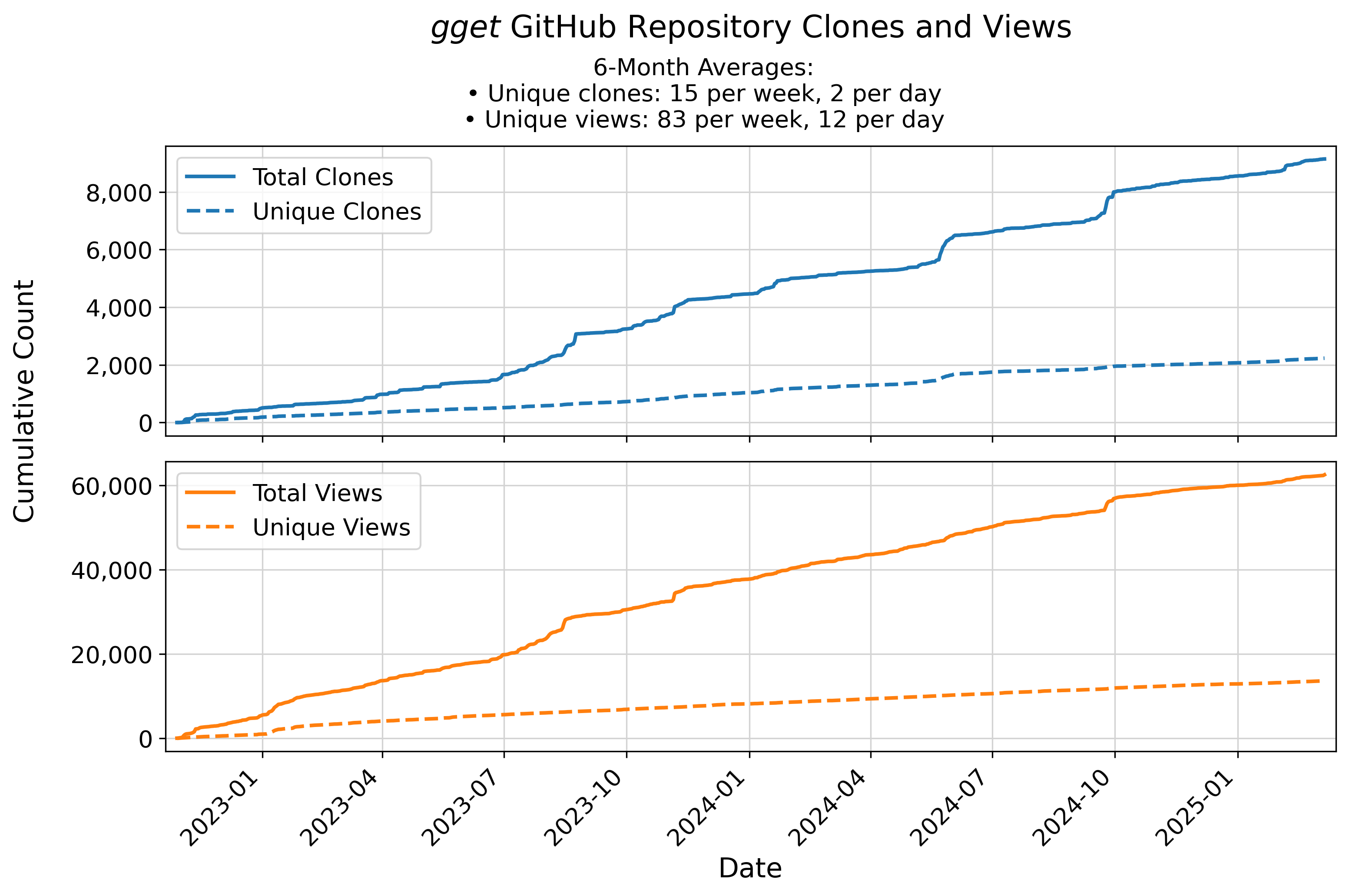
🚂 Tráfico del repositorio de gget