Ver el codigo fuente de la pagina en GitHub
Parámetros de Python són iguales a los parámetros largos (
--parámetro) de Terminal, si no especificado de otra manera. Las banderas son parámetros de verdadero o falso (True/False) en Python. El manuál para cualquier modulo de gget se puede llamar desde la Terminal con la bandera-h--help.
gget enrichr 💰
Realice un análisis de enriquecimiento de una lista de genes utilizando Enrichr.
Produce: Resultados en formato JSON (Terminal) o Dataframe/CSV (Python).
Parámetro posicional
genes
Lista de nombres cortos (símbolos) de los genes de interés para realizar el análisis de enriquecimiento, p. PHF14 RBM3 MSL1 PHF21A.
Alternativamente: use la bandera --ensembl para ingresar IDs tipo Ensembl, p. ENSG00000106443 ENSG00000102317 ENSG00000188895.
Otros parámetros requeridos
-db --database
Base de datos que será utilizada como referencia para el análisis de enriquecimiento.
Admite cualquier base de datos enumerada aquí o uno de los siguientes accesos directos:
'pathway' (KEGG_2021_Human)
'transcription' (ChEA_2016)
'ontology' (GO_Biological_Process_2021)
'diseases_drugs' (GWAS_Catalog_2019)
'celltypes' (PanglaoDB_Augmented_2021)
'kinase_interactions' (KEA_2015)
Parámetros opcionales
-s --species
Especies a utilizar como referencia para el análisis de enriquecimiento. (Por defecto: human)
Opciones:
| Species | Database list |
|---|---|
human | Enrichr |
mouse | Equivalente al humano |
fly | FlyEnrichr |
yeast | YeastEnrichr |
worm | WormEnrichr |
fish | FishEnrichr |
-bkg_l --background_list
Lista de nombres cortos (símbolos) de genes de 'background' (de fondo/control), p. NSUN3 POLRMT NLRX1.
Alternativamente: usa la bandera --ensembl_background para ingresar IDs tipo Ensembl.
-o --out
Ruta al archivo en el que se guardarán los resultados, p. ruta/al/directorio/resultados.csv (o .json). Por defecto: salida estándar (STDOUT).
Para Python, usa save=True para guardar los resultados en el directorio de trabajo actual.
-ko --kegg_out
Ruta al archivo png en el que se guardará la imágen de la vía de señalización celular KEGG, p. ej. ruta/al/directorio/KEGG.png. (Por defecto: None)
-kr --kegg_rank
Rango de la ruta KEGG que se va a trazar. (Por defecto: 1)
figsize
Solo para Python. (ancho, alto) de la visualización en pulgadas. (Por defecto: (10,10))
ax
Solo para Python. Ingresa un objeto de ejes matplotlib para personalizar la visualización.(Por defecto: None)
Banderas
-e --ensembl
Usa esta bandera si genes se ingresa como una lista de IDs tipo Ensembl.
-e_b --ensembl_bkg
Usa esta bandera si background_list se ingresa como una lista de IDs tipo Ensembl.
-bkg --background
Use un conjunto de 20,625 genes 'background'
listados aquí.
-csv --csv
Solo para Terminal. Produce los resultados en formato CSV.
Para Python, usa json=True produce los resultados en formato JSON.
-q --quiet
Solo para Terminal. Impide la información de progreso de ser exhibida durante la ejecución del programa.
Para Python, usa verbose=False para imipidir la información de progreso de ser exhibida durante la ejecución del programa.
plot
Solo para Python. plot=True provée la visualización de los primeros 15 resultados (por defecto: False).
Ejemplo
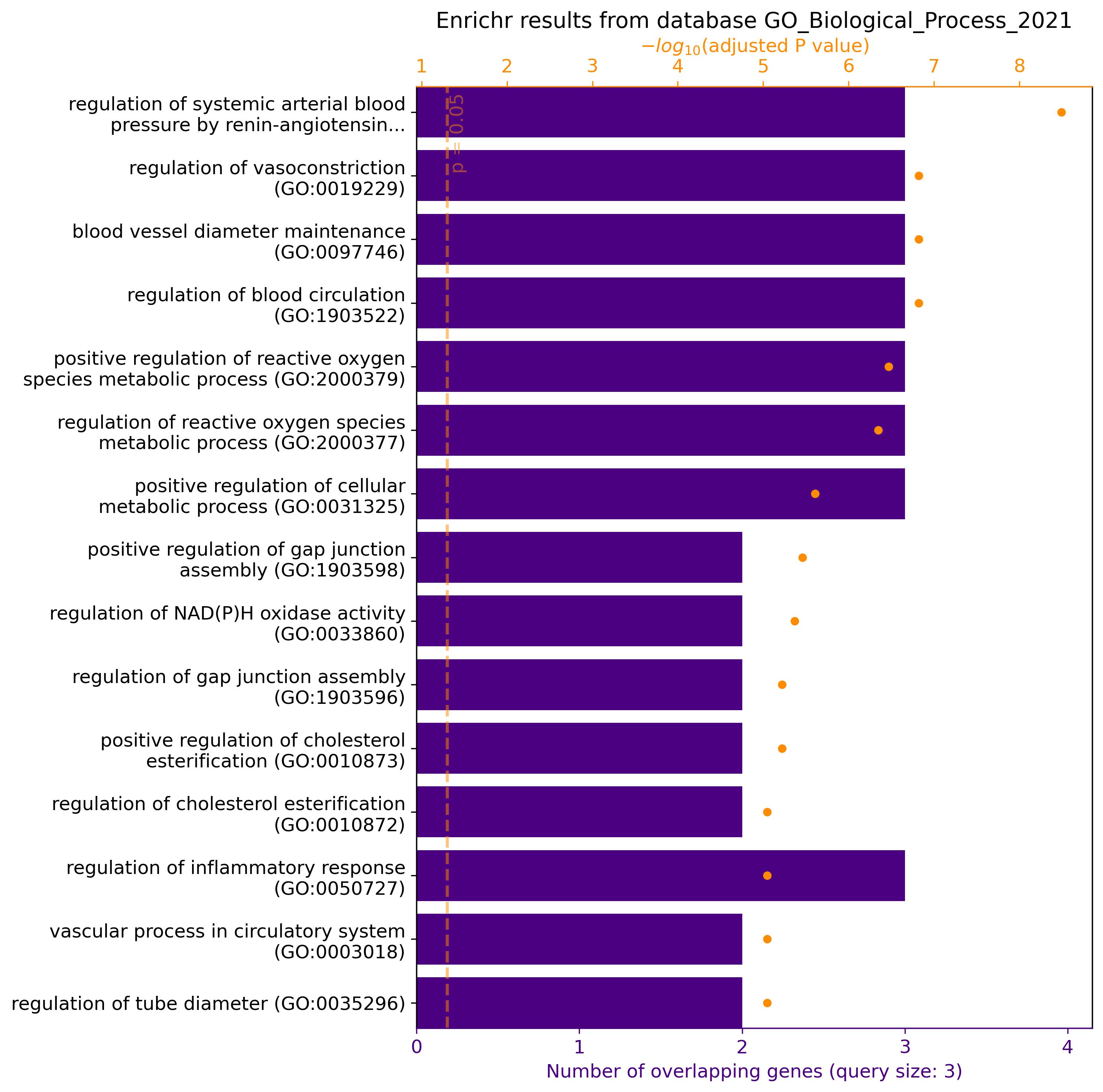
gget enrichr -db ontology ACE2 AGT AGTR1
# Python
gget.enrichr(["ACE2", "AGT", "AGTR1"], database="ontology", plot=True)
→ Produce vías/funciones celulares relacionadas con los genes ACE2, AGT y AGTR1 de la base de datos GO Biological Process 2021. En Python, plot=True provee la visualización de resultados:
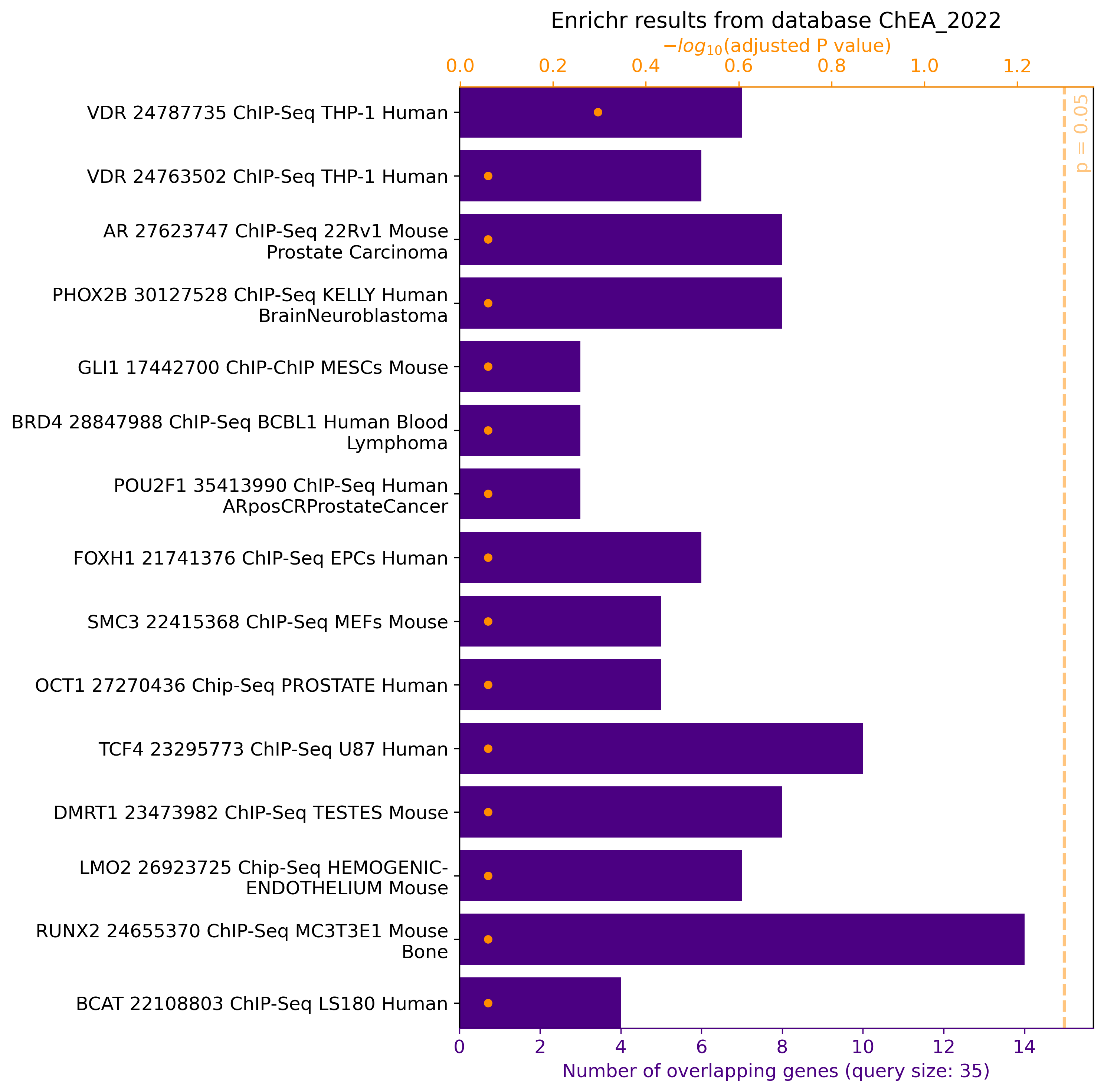
Use gget enrichr con una lista de genes 'background':
# Aquí, primero ingresamos los genes de interés (parámetro posicional 'genes'), para que no se agreguen a la lista de genes 'background' detrás del parámetro '-bkgr_l'
gget enrichr \
PHF14 RBM3 MSL1 PHF21A ARL10 INSR JADE2 P2RX7 LINC00662 CCDC101 PPM1B KANSL1L CRYZL1 ANAPC16 TMCC1 CDH8 RBM11 CNPY2 HSPA1L CUL2 PLBD2 LARP7 TECPR2 ZNF302 CUX1 MOB2 CYTH2 SEC22C EIF4E3 ROBO2 ADAMTS9-AS2 CXXC1 LINC01314 ATF7 ATP5F1 \
-db ChEA_2022 \
-bkg_l NSUN3 POLRMT NLRX1 SFXN5 ZC3H12C SLC25A39 ARSG DEFB29 PCMTD2 ACAA1A LRRC1 2810432D09RIK SEPHS2 SAC3D1 TMLHE LOC623451 TSR2 PLEKHA7 GYS2 ARHGEF12 HIBCH LYRM2 ZBTB44 ENTPD5 RAB11FIP2 LIPT1 INTU ANXA13 KLF12 SAT2 GAL3ST2 VAMP8 FKBPL AQP11 TRAP1 PMPCB TM7SF3 RBM39 BRI3 KDR ZFP748 NAP1L1 DHRS1 LRRC56 WDR20A STXBP2 KLF1 UFC1 CCDC16 9230114K14RIK RWDD3 2610528K11RIK ACO1 CABLES1 LOC100047214 YARS2 LYPLA1 KALRN GYK ZFP787 ZFP655 RABEPK ZFP650 4732466D17RIK EXOSC4 WDR42A GPHN 2610528J11RIK 1110003E01RIK MDH1 1200014M14RIK AW209491 MUT 1700123L14RIK 2610036D13RIK PHF14 RBM3 MSL1 PHF21A ARL10 INSR JADE2 P2RX7 LINC00662 CCDC101 PPM1B KANSL1L CRYZL1 ANAPC16 TMCC1 CDH8 RBM11 CNPY2 HSPA1L CUL2 PLBD2 LARP7 TECPR2 ZNF302 CUX1 MOB2 CYTH2 SEC22C EIF4E3 ROBO2 ADAMTS9-AS2 CXXC1 LINC01314 ATF7 ATP5F1COX15 TMEM30A NSMCE4A TM2D2 RHBDD3 ATXN2 NFS1 3110001I20RIK BC038156 C330002I19RIK ZFYVE20 POLI TOMM70A LOC100047782 2410012H22RIK RILP A230062G08RIK PTTG1IP RAB1 AFAP1L1 LYRM5 2310026E23RIK SLC7A6OS MAT2B 4932438A13RIK LRRC8A SMO NUPL2
# Python
gget.enrichr(
genes = [
"PHF14", "RBM3", "MSL1", "PHF21A", "ARL10", "INSR", "JADE2", "P2RX7",
"LINC00662", "CCDC101", "PPM1B", "KANSL1L", "CRYZL1", "ANAPC16", "TMCC1",
"CDH8", "RBM11", "CNPY2", "HSPA1L", "CUL2", "PLBD2", "LARP7", "TECPR2",
"ZNF302", "CUX1", "MOB2", "CYTH2", "SEC22C", "EIF4E3", "ROBO2",
"ADAMTS9-AS2", "CXXC1", "LINC01314", "ATF7", "ATP5F1"
],
database = "ChEA_2022",
background_list = [
"NSUN3","POLRMT","NLRX1","SFXN5","ZC3H12C","SLC25A39","ARSG",
"DEFB29","PCMTD2","ACAA1A","LRRC1","2810432D09RIK","SEPHS2",
"SAC3D1","TMLHE","LOC623451","TSR2","PLEKHA7","GYS2","ARHGEF12",
"HIBCH","LYRM2","ZBTB44","ENTPD5","RAB11FIP2","LIPT1",
"INTU","ANXA13","KLF12","SAT2","GAL3ST2","VAMP8","FKBPL",
"AQP11","TRAP1","PMPCB","TM7SF3","RBM39","BRI3","KDR","ZFP748",
"NAP1L1","DHRS1","LRRC56","WDR20A","STXBP2","KLF1","UFC1",
"CCDC16","9230114K14RIK","RWDD3","2610528K11RIK","ACO1",
"CABLES1", "LOC100047214","YARS2","LYPLA1","KALRN","GYK",
"ZFP787","ZFP655","RABEPK","ZFP650","4732466D17RIK","EXOSC4",
"WDR42A","GPHN","2610528J11RIK","1110003E01RIK","MDH1","1200014M14RIK",
"AW209491","MUT","1700123L14RIK","2610036D13RIK",
"PHF14", "RBM3", "MSL1", "PHF21A", "ARL10", "INSR", "JADE2",
"P2RX7", "LINC00662", "CCDC101", "PPM1B", "KANSL1L", "CRYZL1",
"ANAPC16", "TMCC1","CDH8", "RBM11", "CNPY2", "HSPA1L", "CUL2",
"PLBD2", "LARP7", "TECPR2", "ZNF302", "CUX1", "MOB2", "CYTH2",
"SEC22C", "EIF4E3", "ROBO2", "ADAMTS9-AS2", "CXXC1", "LINC01314", "ATF7",
"ATP5F1""COX15","TMEM30A","NSMCE4A","TM2D2","RHBDD3","ATXN2","NFS1",
"3110001I20RIK","BC038156","C330002I19RIK","ZFYVE20","POLI","TOMM70A",
"LOC100047782","2410012H22RIK","RILP","A230062G08RIK",
"PTTG1IP","RAB1","AFAP1L1", "LYRM5","2310026E23RIK",
"SLC7A6OS","MAT2B","4932438A13RIK","LRRC8A","SMO","NUPL2"
],
plot=True
)
→ Provée factores de transcripción relacionados a los genes de interés y controlados con la lista de genes background de la base de datos ChEA 2022. En Python, plot=True permite la visualización de resultados:
Genere una imagen de la vía de señalización de células KEGG con los genes del análisis de enriquecimiento resaltados:
Esta función está disponible gracias a un PR de Noriaki Sato.
gget enrichr -db pathway --kegg_out kegg.png --kegg_rank 1 ZBP1 IRF3 RIPK1
# Python
gget.enrichr(["ZBP1", "IRF3", "RIPK1"], database="pathway", kegg_out="kegg.png", kegg_rank=1)
→ Además de los resultados estándar gget enrichr, el argumento kegg_out guarda una imagen con los genes del análisis de enriquecimiento resaltados guardado como kegg.png:
El siguiente ejemplo fue enviado por Dylan Lawless a través de un PR (con ajustes de Laura Luebbert):
Use gget enrichr en R y cree unq visualización similar usando ggplot.
TENGA EN CUENTA el cambio de ejes en comparación con la visualización en Python.
system("pip install gget")
install.packages("reticulate")
library(reticulate)
gget <- import("gget")
# Perform enrichment analysis on a list of genes
df <- gget$enrichr(list("ACE2", "AGT", "AGTR1"), database = "ontology")
# Count number of overlapping genes
df$overlapping_genes_count <- lapply(df$overlapping_genes, length) |> as.numeric()
# Only keep the top 15 results
df <- df[1:15, ]
# Plot
library(ggplot2)
df |>
ggplot() +
geom_bar(aes(
x = -log10(adj_p_val),
y = reorder(path_name, -adj_p_val)
),
stat = "identity",
fill = "lightgrey",
width = 0.5,
color = "black") +
geom_text(
aes(
y = path_name,
x = (-log10(adj_p_val)),
label = overlapping_genes_count
),
nudge_x = 0.75,
show.legend = NA,
color = "red"
) +
geom_text(
aes(
y = Inf,
x = Inf,
hjust = 1,
vjust = 1,
label = "# of overlapping genes"
),
show.legend = NA,
color = "red"
) +
geom_vline(linetype = "dotted", linewidth = 1, xintercept = -log10(0.05)) +
ylab("Pathway name") +
xlab("-log10(adjusted P value)")
Más ejemplos
Citar
Si utiliza gget enrichr en una publicación, favor de citar los siguientes artículos:
-
Luebbert, L., & Pachter, L. (2023). Efficient querying of genomic reference databases with gget. Bioinformatics. https://doi.org/10.1093/bioinformatics/btac836
-
Chen EY, Tan CM, Kou Y, Duan Q, Wang Z, Meirelles GV, Clark NR, Ma'ayan A. Enrichr: interactive and collaborative HTML5 gene list enrichment analysis tool. BMC Bioinformatics. 2013; 128(14). https://doi.org/10.1186/1471-2105-14-128
-
Kuleshov MV, Jones MR, Rouillard AD, Fernandez NF, Duan Q, Wang Z, Koplev S, Jenkins SL, Jagodnik KM, Lachmann A, McDermott MG, Monteiro CD, Gundersen GW, Ma'ayan A. Enrichr: a comprehensive gene set enrichment analysis web server 2016 update. Nucleic Acids Research. 2016; gkw377. doi: 10.1093/nar/gkw377
-
Xie Z, Bailey A, Kuleshov MV, Clarke DJB., Evangelista JE, Jenkins SL, Lachmann A, Wojciechowicz ML, Kropiwnicki E, Jagodnik KM, Jeon M, & Ma’ayan A. Gene set knowledge discovery with Enrichr. Current Protocols, 1, e90. 2021. doi: 10.1002/cpz1.90.
Si trabaja con conjuntos de datos no humanos/ratón, cite también:
- Kuleshov MV, Diaz JEL, Flamholz ZN, Keenan AB, Lachmann A, Wojciechowicz ML, Cagan RL, Ma'ayan A. modEnrichr: a suite of gene set enrichment analysis tools for model organisms. Nucleic Acids Res. 2019 Jul 2;47(W1):W183-W190. doi: 10.1093/nar/gkz347. PMID: 31069376; PMCID: PMC6602483.